|
|
Mécanismes nucléaires de contrôle-qualité des ARN eucaryotes |
|
L’accumulation physiologique de macromolécules anormales peut, au cours du
temps, devenir dommageable à la survie cellulaire ; leur élimination est par
conséquent essentielle. Le métabolisme de l’ARN produit une grande diversité de
substrats à dégrader, comme des sous-produits d’épissage (introns) ou de
maturation des ARNr, des ARN messagers (ARNm) non fonctionnels, des ARN non codants mal
repliés, ou encore des transcrits en fin de vie. La dégradation de ces ARN constitue
un mécanisme d’ébouage cellulaire, éliminant des transcrits inutiles
et encombrants, voire potentiellement toxiques s’ils entrent dans les voies métaboliques mais
ne sont pas fonctionnels. De plus, les nucléotides issus de la dégradation peuvent
être récyclés pour synthétiser de nouveaux acides nucléiques.
Plusieurs mécanismes distincts sont impliqués dans la
dégradation de ces espèces, qui s’effectue majoritairement par digestion
exonucléolytique, soit de l’extrémité 5’ vers l’extrémité 3’, soit
de 3’ vers 5’ (pour revue, voir Isken et Maquat, Genes Dev., 2007).
Un complexe multi-protéique appelé exosome assure la majeure partie des dégradations 3’—5’ (pour
revue, Lebreton et
Séraphin, Biochim. Biophys. Acta 2008; Siwaszek et al., RNA Biol. 2014). Il est
constitué d’un cœur de 10 sous-unités (Figure 1) ; neuf d’entre elles forment une structure en
anneau apparentée à la PNPase bactérienne ou à l’exosome d’archées (Liu et al.,
Cell 2006), mais dépourvue d’activité ; la dixième sous-unité, Dis3/Rrp44, possède
un site RNase II exerçant une activité exoribonucléolytique 3’—5’ (Dziembowski et al.,
Nat. Struct. Mol. Biol. 2007). Dans le noyau, une autre RNase 3’—5’, Rrp6,
s’associe à ce complexe avec son cofacteur Rrp47.
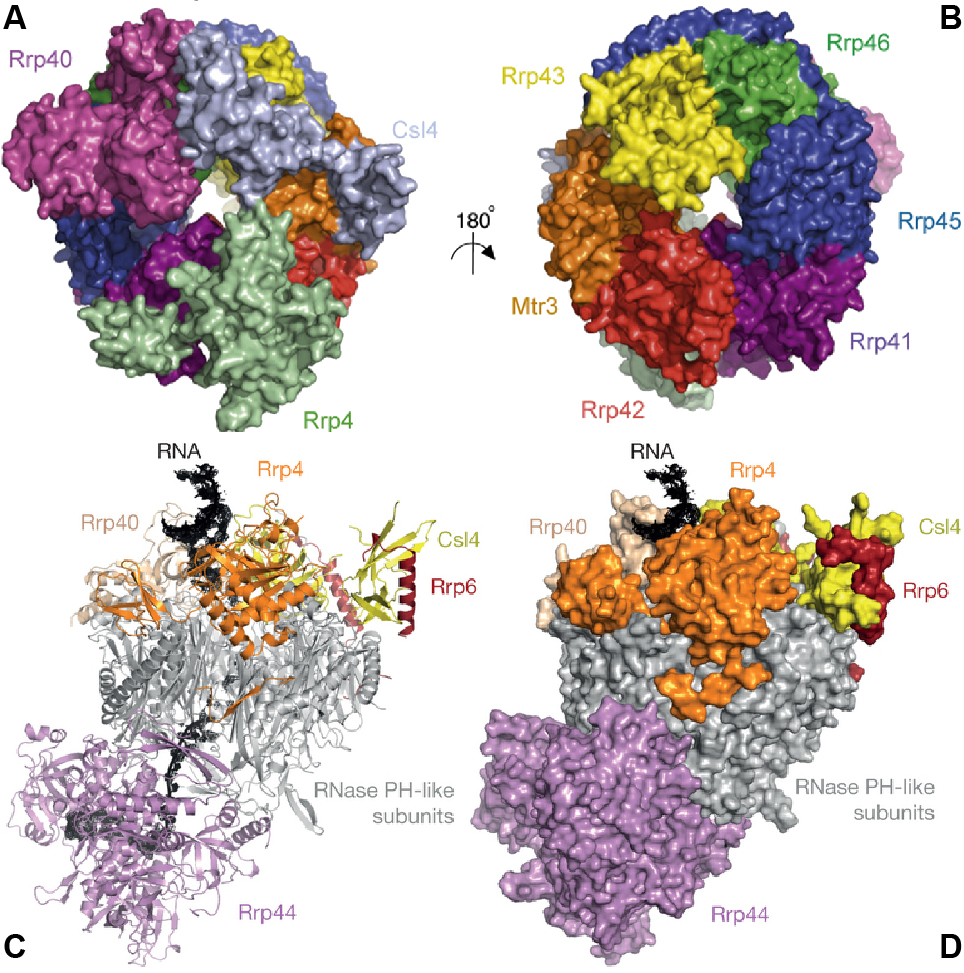
Figure 1. Structure de l’exosome eucaryote. Les neuf sous-unités de la structure en anneau sont
représentées en vue de dessus (A) ou de dessous (B) ; elles ménagent un
canal central au creux duquel l’ARN peut passer (Liu et al., Cell, 2006).
(C-D) Le site d’interaction de Dis3/Rrp44 est positionné sur la face inférieure,
à la sortie du canal qui achemine le substrat ARN depuis la face supérieure
(Makino et al., Nature 2013). Le dimère Rrp6-Rrp47 vient se positionner
sur la face supérieure, à l’entrée du canal d’accès (seule une partie de Rrp6 apparaît ici).
Dans le cytoplasme, l’activité de l’exosome contribue à la “voie de dégradation mineure” des ARNm
(Anderson et Parker, EMBO J. 1998), participe au non-stop decay (NSD), un processus de dégradation
des ARNm dépourvus de codon stop (van Hoof et al. Science 2002) et au nonsense-mediated decay (NMD),
qui élimine les ARNm portant un codon stop précoce (Mitchell et Tollervey, Mol. Cell 2003). Dans le
noyau, l’exosome est requis pour la maturation des ARNr, ainsi que des petits ARN nucléaires et
nucléolaires (Allmang et al., EMBO J. 1999), et constitue le principal acteur des mécanismes de
contrôle-qualité qui s’exercent sur les transcrits naissants.
Sous la bannière d’un mécanisme général, plusieurs voies distinctes coexistent
pour acheminer vers l’exosome ses divers types de substrats (Lebreton et Séraphin, Biochim. Biophys. Acta 2008). De nombreux cofacteurs sont
connus pour leurs fonctions activatrices, et chacun d’entre eux présente un spectre de substrats
caractéristique. Par exemple chez Saccharomyces cerevisiae, les acteurs du contrôle-qualité nucléaire
des ARN orientent certains transcrits aberrants vers une dégradation 3’—5’ par l’exosome
(Houseley et al., Nat. Rev. Mol. Cell. Biol. 2006; Schmidt et Butler, Wiley Interdiscip. Rev. RNA 2013).
Une part de ces transcrits correspond à des ARN dont la maturation est anormale. Ce sont
principalement des ARN de transfert, des petits ARN nucléaires et nucléolaires mal
repliés, ainsi que des précurseurs d’ARNr aberrants.
Une autre part correspond à une catégorie d’ARN instables appelés CUT
(Cryptic Unstable Transcripts). Il s’agit d’ARN produits dans les régions intergéniques
du génome à partir de promoteurs cryptiques, n’ayant pas de site consensuel de terminaison de
transcription et de poly-adénylation par Pap1, ne codant aucun produit polypeptidique. Leur
principale vocation connue à ce jour semble d’être dégradés, et l’essentiel de leurs fonctions
reste en conséquence mal caractérisé ; cependant, ils pourraient intervenir dans certaines régulations de
l’expression génique (Colin et al., Genet. Res. Int. 2011).
La reconnaissance spécifique de ces ARN fait intervenir un complexe de
polyadénylation appelé TRAMP (Trf4/5-Air1/2-Mtr4 Polyadenylation complex). Trf4 et Trf5
appartiennent à une nouvelle famille de poly(A) polymérases non canoniques ; les
queues poly(A) ajoutées par ces enzymes sont plus courtes que celles synthétisées
par Pap1 en 3’ des ARNm, et n’ont pas d’effet stabilisateur ; au contraire, les substrats
polyadénylés par TRAMP sont ensuite pris en charge par l’exosome nucléaire, qui
assure leur dégradation (Figure 2).

Figure 2. Modèle de recrutement du complexe TRAMP et de l’exosome sur les
transcrits aberrants. Les ARN anormaux sont rapidement détectés,
polyadénylés par Trf4 ou Trf5, puis dégradés par les RNases de
l’exosome à partir de l’extrémité 3’.
Outre Trf4 ou Trf5, le complexe TRAMP de levure est constitué des protéines de liaison à
l’ARN Air1
ou Air2 et de Mtr4, protéine de la famille des hélicases /em>à boîte DExD/H, qui participe entre autres
à la maturation des ARNr (la Cruz et al., EMBO J. 1998). Mtr4 interagit également avec l’exosome
nucléaire, et constitue ainsi une interface protéique entre les complexes impliqués dans la
reconnaissance et la dégradation des substrats.
Au cours de mon post-doctorat, j’ai développé plusieurs projets :
j’ai cherché à apporter une meilleure connaissance du rôle de Mtr4 dans
la voie TRAMP chez S. cerevisiae, et à caractériser une machinerie de dégradation
similaire chez les eucaryotes supérieurs. Enfin, ma principale contribution
au domaine a été l’identification d’une activité endonucléolytique
dans l’exosome de levure.
|
|
| Rôle
de l’hélicase Mtr4 dans la voie TRAMP-exosome chez Saccharomyces cerevisiae |
|
Un premier volet concernait l’étude du rôle crucial de l’hélicase Mtr4 dans
l’activité de l’exosome, quelle que soit la voie de recrutement (dépendante ou non de TRAMP).
Plusieurs hypothèses étaient envisagées : Une activité ARN-hélicase de Mtr4 sur les structures
secondaires des ARN facilitant l’action de l’exosome ; une participation directe au recrutement
direct de l’exosome via des interactions protéine-protéine ; et/ou une fonction de translocase
permettant le déplacement de des cofacteurs protéiques du substrat ARN lors de la dégradation par
l’exosome.
Ces travaux n’ont pas été publiés. Un autre groupe a depuis
montré que les activités ATPase et de fusion des duplexes ARN-ARN sont requises pour la fonction de
Mtr4, et qu’elle intervient après la polyadénylation des substrats par Trf4, et avant leur
dégradation par l’exosome (Wang et al., RNA 2008b). Le même groupe a ensuite confirmé
le rôle crucial de Mtr4 dans le contrôle de l’activité du complexe TRAMP (Jia et al.,
Cell 2011), nécessitant entre autres le déroulement des substrats ARN (Jia et al.,
PNAS 2012).
|
|
| Caractérisation
de l’homologue humain de TRAMP |
|
Une seconde partie concernait la caractérisation de l’homologue humain de TRAMP.
Si l’étude de la machinerie nucléaire de dégradation des ARN aberrants était bien avancée chez la
levure, aucun mécanisme analogue n’avait été décrit chez les eucaryotes supérieurs. Cependant, des
intermédiaires polyadénylés d’ARNr anormaux et de pré-ARNm tronqués avaient été identifiés dans les
cellules humaines (Slomovic et al., Nucleic Acids Res. 2006; West et al., Mol. Cell 2006). Ceci
laissait penser qu’une activité similaire à celle de TRAMP existait dans les cellules humaines et
pouvait, comme chez la levure, orienter les ARN aberrants vers une dégradation par l’exosome.
Ce dernier est également retrouvé chez les eucaryotes supérieurs (Sloan et al., Biochem. Soc. Trans.
2012). Par
similitude de séquence, nous avons identifié des homologues humains des constituants de TRAMP,
suggérant que la machinerie de polyadénylation nucléaire des ARN aberrants était conservée.
Avec l’aide d’une assistante-ingénieure, Marie-élisabeth Dufour, nous
avons cloné et purifié les homologues humains des protéines du complexe et montré leur interaction
in vitro. L’un des complexes ainsi formés présentait une activité de polyadénylation sur des ARN
transcrits in vitro, et stimulait la dégradation d’un substrat par l’exosome de levure. Enfin, tous
les candidats étudiés avaient une localisation majoritairement nucléaire (Figure 3, non publié).
Le complexe
TRAMP humain a depuis été caractérisé par d’autres groupes (Fasken et al., J. Biol. Chem. 2011;
Lubas et al., Mol. Cell 2011).
|
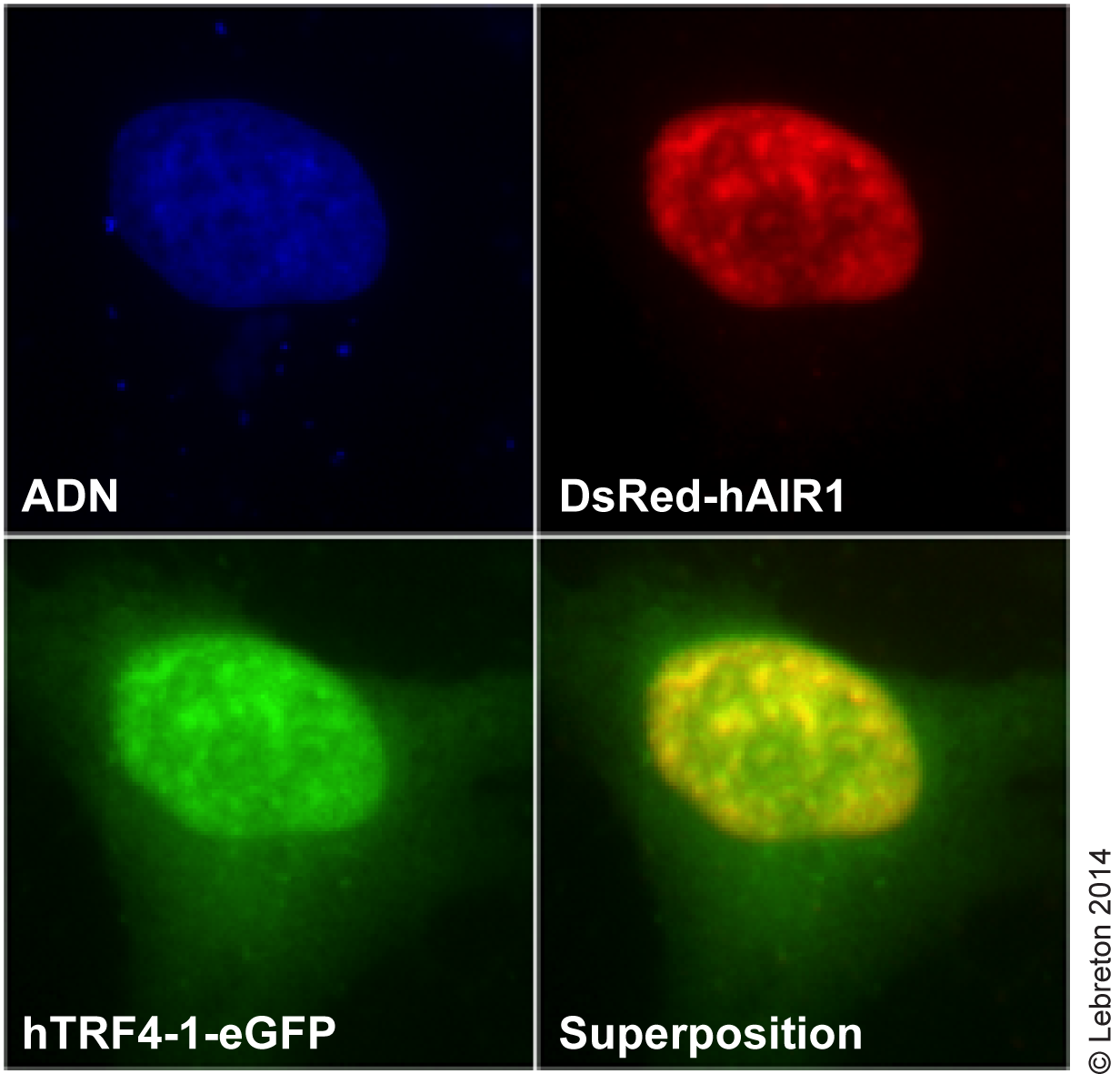
Figure 3. Co-localisation nucléaire de deux constituants du complexe TRAMP humain.
|
|
| Activité
endonucléolytique de l’exosome eucaryote |
|
L’exonucléase principale Dis3 possède, en plus de son principal
domaine catalytique, un domaine PIN, essentiel et de fonction inconnue (Figure 4). Afin de mieux
cerner son rôle, j’ai construit différents mutants ponctuels ciblant des acides
aminés très conservés dans les différentes régions de Dis3.
Les protéines mutées sont incorporées sont produites et incorporées à la structure de l’exosome
de levure avec la même efficacité que la protéine sauvage.
Ces mutants ne complémentent toutefois pas le
défaut de croissance observé en l’absence de Dis3, indiquant un rôle
physiologique des domaines ciblés par la mutagenèse.
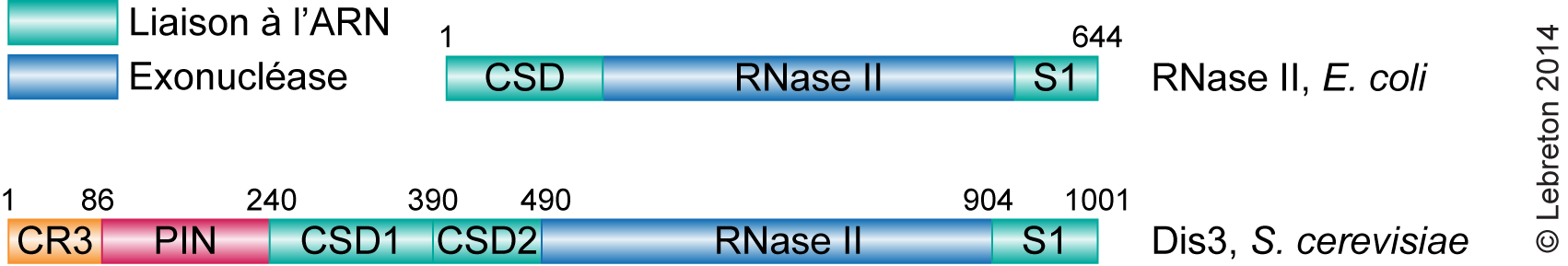
Figure 4. Répartition des différents domaines fonctionnels le long de la séquence
de Dis3, comparée à la RNase II procaryote. CR3, domaine de fonction inconnue,
contenant 3 résidus cystéine ; PIN, homologie au domaine N-terminal de PilT (activité endonucléase) ;
CSD, Cold Shock Domain (OB-fold) ; S1, similaire au domaine de liaison à l’ADN de la protéine
ribosomique S1 (OB-fold). Les deux domaines CR3 et PIN sont
spécifiques à la protéine eucaryote.

Lors de l’expression d’un mutant du domaine
RNase II dépourvu d’activité exonucléolytique, de nouveaux sites de clivage dans certains substrats de Dis3, en
particulier des CUT et des précurseurs d’ARNr (Figure 5). Ces sites de clivage disparaissent chez un double
mutant du domaine PIN et du site exonucléase ; le transcrit précurseur s’accumule
alors fortement. Le domaine PIN exerce par conséquent un rôle dans la dégradation
de certains ARN, indépendamment de l’activité exonucléolytique de Dis3
(Lebreton, Tomecki et al.,
Nature, 2008).
J’ai caractérisé par extension d’amorces et circular-RACE
(circularisation puis RT-PCR) les intermédiaires accumulés par le mutant du domaine
RNase II pour l’un des substrats de Dis3. Ils recouvrent l’ensemble de la séquence du
transcrit précurseur, et possèdent des extrémités 5’ et 3’ bien
définies, localisées dans des régions non appariées. Ces données
in vivo nous ont permis de déduire que le domaine PIN exerce une fonction d’endoribonucléase
simple brin (Lebreton, Tomecki et al.,
Nature, 2008). Par ailleurs, des travaux réalisés in vitro par nos
collaborateurs R. Tomecki et A. Dziembowski (Département de Génétique et
Biotechnologies, Université de Varsovie, Pologne) ont confirmé l’existence de cette
activité. Deux autres groupes sont peu après parvenus à des conclusions similaires
(Schaeffer et al., Nat. Struct. Mol. Biol. 2009; Schneider et al., Nucleic Acids Res. 2009).
La protéine Dis3 de levure est ainsi devenue la premier représentante de protéines
portant les deux activités, exonucléase et endonucléase, dans des domaines distincts.
Ces résultats se sont également avérés critiques pour les travaux ultérieurs de caractérisation
structurale et fonctionnelle de l’exosome. En effet, l’obtention d’une version mutante de l’exosome
totalement dépourvue d’activité ribonucléolytique était un prérequis pour établir la structure du complexe
avec un substrat ARN, et ainsi cartographier les canaux moléculaires acheminant l’ARN vers les sites actifs
(Makino et al., Nature. 2013).
Figure 5. Effets des mutants de Dis3 sur la dégradation des substrats de
l’exosome. Exo- désigne le mutant du domaine RNase II, Endo- le mutant du domaine PIN.
|
|
|
Caractérisation des substrats physiologiques de l’exosome |
|
En observant l’incidence de mutations touchant Rrp6 ou Dis3 sur différents
substrats (précurseurs d’ARNr, CUT et snARN), j’avais constaté, malgré la redondance fonctionnelle des
deux nucléases, une préférence relative pour certains types de transcrits. Ces données laissaient
penser que les substrats pourraient être orientés au sein de l’exosome vers différentes voies de
dégradation, prédéfinies en amont par des cofacteurs comme TRAMP.
Afin de mieux comprendre les mécanismes coopératifs de dégradation des ARN et de leur rôle physiologique,
les équipes d’Alain Jacquier et Domenico Libri ont poursuivi l’étude
de la contribution des différentes activités endo- et exo-nucléolytiques de l’exosome à
l’élimination des précurseurs d’ARN
(Gudipati et al., Mol. Cell 2012).
Cette étude a confirmé la redondance
fonctionnelle partielle de Dis3 et Rrp6 pour la dégradation de différentes classes de substrats,
comme par exemple des transcrits immatures avant leur épissage. De façon surprenante, une grande
quantité de précurseurs d’ARNt est également dégradée en l’absence de tout défaut de maturation.
Ceci suggère que la machinerie de dégradation entre en compétition cinétique avec les voies de
maturation des ARN cellulaires, permettant une surveillance de l’efficacité de la maturation, mais
conduisant également à l’élimination précoce de nombreux transcrits.
|
|
|
Conclusion |
|
Depuis la découverte de l'exosome eucaryote, les recherches réalisées par
plusieurs équipes ont conduit à revoir radicalement son mode d’action.
En effet, les premières études laissaient penser qu’il s’agissait d’un complexe
constitué d’une dizaine d’exonucléases 3’—5’ (Mitchell et al., Cell, 1997).
Les études structurales et fonctionnelles (Dziembowski et al., Nat.
Struct. Mol. Biol., 2007; Liu et al., Cell — erratum, 2007) ont en revanche montré que
neuf des sous-unités du cœur de l’exosome sont dépourvues d’activité,
et que seules Dis3 et Rrp6 sont des exonucléases.
Mes travaux se sont inscrits dans la continuité de ces études, en
révélant l’existence d’un domaine endonucléase au sein de Dis3. Ce domaine
pourrait assister les fonctions exonucléolytiques de l’exosome, par exemple en créant des
extrémités supplémentaires dans les substrats et ainsi permettre une
réinitiation de la dégradation lorsque la progression de l’exosome est bloquée
(Fig. 6).
Aussi bien chez la levure que chez l’homme, la fonction des neuf
sous-unités formant la structure en anneau de l’exosome est encore incertaine. Les
hypothèses actuelles sont en faveur d’un rôle de cette structure dans la
régulation des activités des nucléases, en permettant un accès
sélectif des substrats aux sites catalytiques via le canal central, et en favorisant l’ancrage des
différents cofacteurs de l’exosome qui assistent la dégradation.
|

Figure 6. Modèle envisagé pour l’implication de l’activité
endonucléolytique du domaine PIN de Dis3 dans la dégradation des substrats de
l’exosome.
|
|
|
|





