|
|
La biogenèse des ribosomes chez Saccharomyces cerevisiae |
|
Le processus d’assemblage des ribosomes est très conservé, étroitement régulé en réponse aux stress
environnementaux et aux conditions de croissance cellulaire. Il consomme, dans les cellules eucaryotes en
prolifération, plus de 50% de l’énergie produite. Cette voie métabolique débute dans le nucléole, se poursuit
dans le nucléoplasme et s’achève dans le cytoplasme (Figure 1).

Figure 1. Un aperçu de la biogenèse des ribosomes chez
Saccharomyces cerevisiae. Les ARN polymérases I et III transcrivent le précurseur d’ARN
ribosomique (ARNr) 35S et l’ARNr 5S, qui s’associent dans le nucléole aux protéines ribosomiques et
aux facteurs pré-ribosomiques pour former la particule 90S. La biogenèse suit ensuite deux voies
distinctes, la pré-60S qui produira la grande sous-unité ribosomique, et la pré-40S qui conduira à
la petite. Une séquence ordonnée de réactions de maturation des ARNr, conjointe à l’association et
à la dissociation de facteurs protéiques, accompagne le transport des particules vers le
nucléoplasme, puis le cytoplasme, où deux sous-unités matures s’associent à un ARN messager (ARNm)
pour former un ribosome 80S lors de la traduction.
Les sous-unités ribosomiques 40S et 60S sont des structures
ribonucléoprotéiques complexes : les protéines ribosomiques s’associent
à deux transcrits précurseurs, le 5S et le 35S. Ce dernier est maturé par
élimination de séquences espaceurs externes (ETS) et internes (ITS), pour donner les
ARN ribosomiques (ARNr) matures 18S, 5,8S et 25S (Figure 2).

Figure 2. Principaux sites de clivage du précurseur 35S lors de la maturation des ARNr 18S, 5,8S et 25S.
La biogenèse nécessite l’intervention d’un grand nombre de facteurs,
dits «pré-ribosomiques», spécifiques aux cellules eucaryotes. En effet, chez les
procaryotes, les ARNr matures et les protéines ribosomiques peuvent s’auto-assembler in
vitro, en présence d’une source de chaleur, pour former des ribosomes fonctionnels
(Traub et Nomura, J. Mol. Biol., 1969). Chez les eucaryotes, la compartimentation, ainsi que
l’importance accrue du rôle des protéines ribosomiques dans la structure finale, ont
conduit à une complexification du processus d’assemblage. Depuis le début des
années 2000, de nombreux travaux ont permis de mieux cerner la composition des particules
pré-ribosomiques eucaryotes (Nissan et al., EMBO J., 2002; Krogan et al., Mol.
Cell, 2004; Gavin et al., Nature, 2006). L’analyse fonctionnelle du rôle de chacun
des facteurs, de son ordre d’intervention et de leur inter-dépendance se poursuit (pour revues voir
Fromont-Racine et al., Gene, 2003,
Gerhardy et al., Chromosoma2014).
|
|
|
Dynamique des facteurs pré-ribosomiques au cours de la biogenèse de la
grande sous-unité ribosomique chez Saccharomyces cerevisiae |
|
Les facteurs pré-ribosomiques ne participent que temporairement à
la biogenèse, et ne se retrouvent pas dans l’architecture finale des ribosomes matures. Ils
exercent leurs fonctions catalytiques, régulatrices ou de transport des particules selon un
ordre bien précis, puis sont libérés, pour être potentiellement
recyclés. Nous avons montré que l’ordre d’assemblage des protéines sur les
particules pré-60S s’opérait de manière séquentielle et reposait, au
moins en partie, sur des interactions protéine-protéine
(Saveanu
et al., Mol. Cell. Biol., 2003).
Les travaux que j’ai réalisés au cours de ma thèse ont permis
la caractérisation fonctionnelle de nouveaux facteurs participant à deux points de
contrôle de la biogenèse de la grande sous-unité ribosomique, l’un
dans le noyau, l’autre dans le cytoplasme. Par l’utilisation de
techniques très diverses (purifications par affinité en tandem, microscopie, cribles
génomiques, reconstitution d’interactions in vitro, protéomique
semi-quantitative, etc.), j’ai cherché à approfondir les aspects dynamiques de la
biogenèse, en essayant de décrypter l’ordre d’assemblage, de
dissociation et de recyclage de ces facteurs pré-60S, tout au long de la voie
métabolique.
Le manuscrit de cette thèse peut être consulté sur le
serveur de thèses en
ligne du Centre pour la Communication Scientifique Directe du CNRS. Vous trouverez ci-dessous un
résumé des principaux résulats obtenus.
|
|
| Nsa2,
un facteur pré-60S extrêmement conservé, impliqué dans un
point clé de la biogenèse des ribosomes : le clivage de l’ITS2 |
|
La protéine nucléaire-nucléolaire Nsa2 a été
identifiée dans plusieurs complexes pré-ribosomiques, et est essentielle pour la
biogenèse de la grande sous-unité ribosomique (Lebreton et al., J. Biol. Chem., 2006). Il s’agit de l’une
des protéines les plus conservées, de la levure aux eucaryotes supérieurs. L’expression de l’orthologue humain,
TINP1 (TGFβ-Inducible Protein 1), est capable de complémenter, bien que très partiellement,
le phénotype de souches de levure où NSA2 est invalidé.
Par des cribles génétiques et des purifications de complexes, j’ai montré que Nsa2 était un partenaire
physique et fonctionnel privilégié de la GTPase Nog1. Toutes deux sont nécessaires pour une étape clé de
l’assemblage de la grande sous-unité ribosomique dans le noyau : le clivage C2, qui permet l’élimination de
l’ITS2 (Internal Transcribed Spacer 2), une séquence d’ARN précurseur
comprise entre les ARNr 5,8S et 25S (Figure 2).
En l’absence de Nsa2, Nog1 est associée à des complexes pré-ribosomiques dont la
maturation est bloquée. En l’absence de Nog1, la biogenèse est aussi bloquée et Nsa2 disparaît des
complexes pré-60S. Plus généralement, la stabilité et donc l’abondance de Nsa2 dépendent de
l’activité de la biogenèse des ribosomes. De fašon intéressante, l’expression de l’orthologue humain
est aussi étroitement régulée ; il est entre-autres surexprimé en réponse au TGFβ.
Depuis 2006, quatre articles ont été publiés concernant le rôle de NSA2/TINP1 humain (Zhang et al.,
Biochem. Biophys. Res. Commun. 2010; Shahni et al., Diabetologia 2012; Int J. Biochem. Cell. Biol. 2013;
Li et al., Oncol. Rep. 2013), principalement dans des journaux de biologie
médicale. Ces études rapportent une localisation nucléolaire de la protéine humaine, identique à son
homologue de levure, et un rôle dans le contrôle de la prolifération cellulaire, comme attendu aussi
bien d’un facteur limitant pour la biogenèse des ribosomes que d’une protéine dont la production est
régulée par le TGFβ. Enfin, l’expression du TGFβ, ainsi que de p21 et p53, est perturbée lorsque
NSA2 est réprimé ou surexprimé. Le rôle de NSA2 humain dans la biogenèse des ribosomes n’a
pas été formellement prouvé toutefois, l’étude de l’homologue humain des protéines
impliquées dans la biogenèse
des ribosomes fait désormais l’objet d’études systématiques, qui devraient dans les prochaines
années permettre de transposer aux eucaryotes supérieurs de nombreux résultats obtenus chez la
levure (Tafforeau et al., Mol. Cell 2013).
|
|
| Définition
de la dynamique d’assemblage des particules 60S par spectrométrie de masse
semi-quantitative sur des complexes purifiés |
|
Si certains facteurs pré-ribosomiques restent associés aux sous-unités en cours de maturation du
nucléole jusqu’au cytoplasme, d’autres au contraire s’associent de manière très transitoire pour accomplir leur
fonction, puis sont décrochés. En raison de cette évolution constante de la composition des particules pré-ribosomiques,
les complexes protéiques purifiés par chromatographie d’affinité en tandem sont en réalité des
mélanges de plusieurs complexes distincts, chacun reflétant une étape précise de la biogenèse. À la suite de nos
travaux sur l’ordre d’assemblage des sous-unités ribosomiques (Saveanu et al., Mol. Cell. Biol., 2003), nous avons souhaité
mieux définir les états successifs de la grande sous-unité ribosomique au cours de sa maturation, en purifiant des
complexes homogènes grâce à l’utilisation de mutants.
Pour ceci, nous avons choisi plusieurs couples de facteurs, dont l’un est utilisé comme appât de
chromatographie d’affinité en tandem, tandis que l’autre est réprimé. Il est ainsi possible d’isoler des sous-complexes
pré-ribosomiques caractéristiques d’une étape donnée de la biogenèse des ribosomes. Ces complexes
sont comparés à ceux purifiés dans un contexte sauvage, par la technique de SILAC (Stable Isotope Labelling
with Amino acids in Cell culture). Cette technique permet des analyses semi-quantitatives par spectrométrie de
masse de la composition protéique des complexes purifiés, gràce à un marquage
différentiel à la leucine deutérée de l’un des échantillons.
Les 6 couples de facteurs utilisés jalonnent la voie de biogenèse de la grande sous-unité. Nous avons
ainsi pu établir des groupes de facteurs intervenant précocement ou tardivement dans la biogenèse, par rapport
au point d’action de chacun des facteurs réprimé (Figure 3). Grâce à ces résultats, nous disposons désormais d’un aperçu
assez complet du scénario d’assemblage de la grande sous-unité ribosomique, du nucléole au cytoplasme
(Lebreton et al., Nucleic Acids Res., 2008).
Ce modèle a été établi d’après les données issues de notre étude et en cohérence avec les travaux
préalablement publiés dans le domaine. Les facteurs indiqués en caractères gras sont ceux ayant servis d’appâts ou de
mutants clés dans notre étude.
|
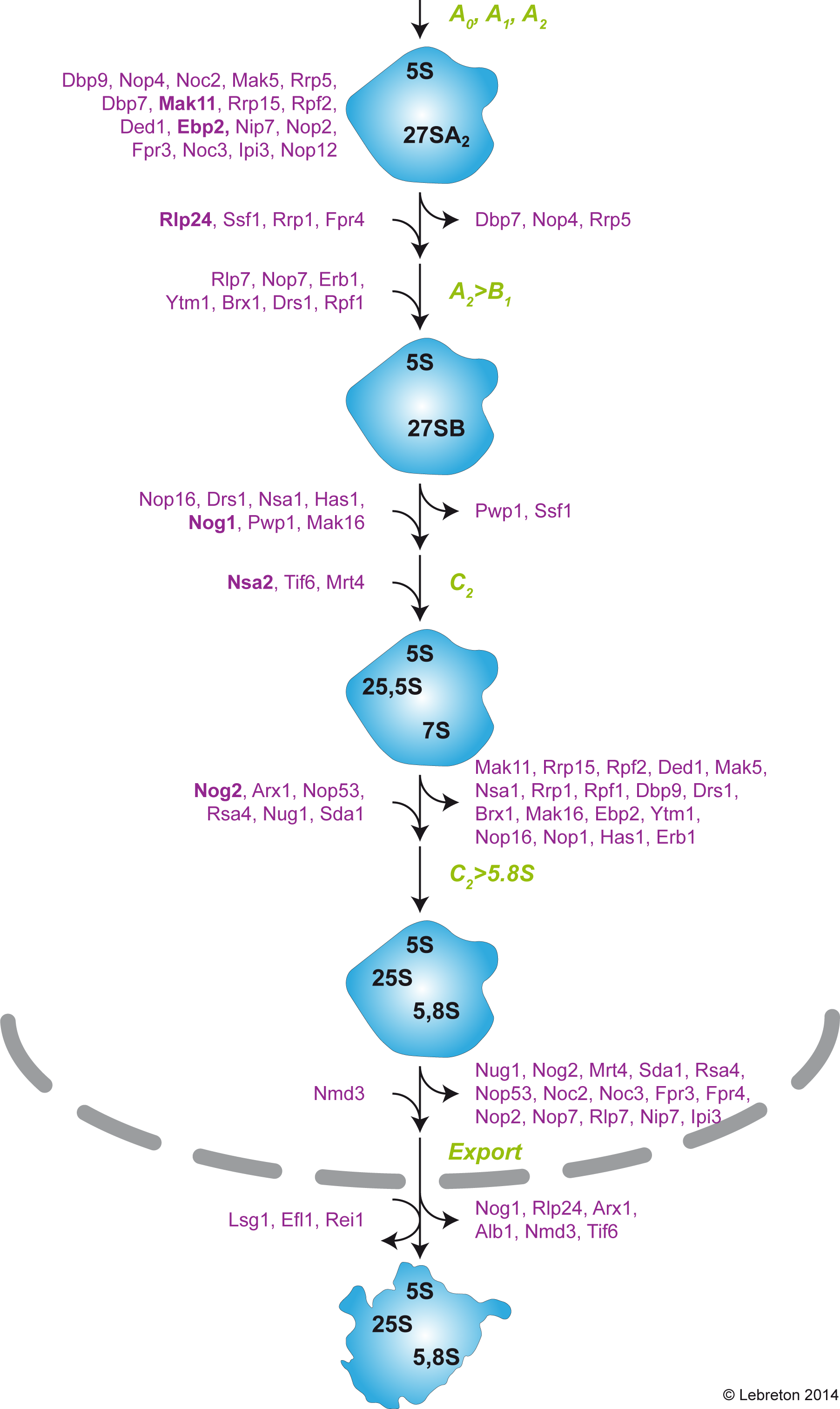
Figure 3. Modèle envisagé pour l’assemblage et la dissociation des facteurs pré-ribosomiques sur les
particules pré-60S.
|
|
| Un
réseau fonctionnel impliqué dans le recyclage des facteurs pré-60S
nucléo-cytoplasmiques |
|
Les étapes finales de fabrication de la grande sous-unité ribosomique sont encore assez mal définies :
dans le cytoplasme, les derniers facteurs pré-ribosomiques, dits «navettes», sont décrochés des particules pré-60S,
et réutilisés pour un nouveau cycle de biogenèse. Quelques protéines exclusivement cytoplasmiques sont
impliquées dans ce processus. Par une combinaison d’approches in vivo et in vitro, j’ai mis en évidence le rôle
crucial d’un réseau d’interactions fonctionnelles, à la transition entre la fin de la biogenèse de la grande sous-unité
ribosomique et l’initiation de la traduction
(Lebreton et al., J. Cell Biol., 2006).
Un nouveau facteur pré-60S cytoplasmique, Ybr267w/Rei1, est lié génétiquement et physiquement à la
protéine ribosomique tardive Rpl24. Paradoxalement, l’absence de ce facteur cytoplasmique se traduit par des
défauts nucléaires de maturation des ARNr. Dans des souches où REI1 est invalidé, l’import nucléaire de trois
facteurs navettes au moins est affecté : Arx1, Tif6 et un nouveau facteur, Yjl122w/Alb1. Arx1 et Alb1 forment
un dimère, dont le retour au noyau dépend d’une β-karyophérine spécifique, Kap121.
En l’absence de Rei1, l’accumulation cytoplasmique du dimère Arx1-Alb1 inhibe le décrochage de Tif6
des particules pré-ribosomiques, et son recyclage vers le noyau. Le déficit nucléaire de Tif6 expliquerait les
défauts de maturation des ARNr observés. Tif6 étant précédemment décrit comme un facteur inhibant
l’association entre la petite et la grande sous-unité ribosomique, nous proposons un
modèle dans lequel Rei1 coordonne la dissociation et le recyclage des derniers facteurs pré-60S, permettant
l’entrée des grandes sous-unités ribosomiques matures dans la traduction (Figure 4).

Figure 4. Contrôle de maturation de la sous-unité 60S à la fin de la biogenèse.
Lors des étapes cytoplasmiques de la biogenèse des particules 60S, le recyclage des facteurs navettes est couplé
au dernier contrôle-qualité de la grande sous-unité ribosomique avant la traduction.
Lors de la publication de nos travaux sur le réseau Rei1-Arx1-Alb1-Tif6, les
données d’un autre groupe sont apparues en contradiction avec nos résultats (Hung et Johnson, Mol. Cell. Biol. 2006).
Nous avions montré qu’en l’absence de Rei1, Arx1 et Alb1 sont dissociées des particules pré-60S et
demeurent dans le cytoplasme sous la forme d’un petit complexe qui inhibe le recyclage de Tif6. Au
contraire, Hung et Johnson étaient parvenus à la conclusion que Rei1 est nécessaire au retour d’Arx1
vers le noyau, mais qu’Arx1 reste associée aux particules pré-60S cytoplasmiques en l’absence de
Rei1.
Je supposais à l’époque dans mon manuscrit de thèse que cette incohérence pouvait provenir,
soit du fond génétique des
souches que nous utilisions, soit d’une différence de salinité des tampons lors de la séparation de
polysomes sur gradients de saccharose. Une réponse à ce problème a été apportée par
une étude très rigoureuse reproduisant les deux résultats (Meyer et al., J. Biol. Chem. 2010) : la différence de
sédimentation s’explique par une concentration en MgCl2 de 30 mM dans notre étude, contre 5 mM pour
l’autre groupe — une concentration plus proche de celle rencontrée dans le cytosol. Cette
explication satisfaisante conduit les auteurs à conclure que Rei1, ainsi que Jjj1, participent à
l’extraction de Arx1 de la sous-unité pré-60S.
La poursuite de la caractérisation de ce réseau d’interactions a donné lieu à la publication d’un travail
en collaboration avec mon ancien laboratoire de thèse et l’équipe d’Helmut Bergler, de l’Institut de Biologie
Moléculaire, à Graz en Autriche (Pertschyet al.,
Mol. Cell. Biol. 2007). Nous avons montré que l’AAA-ATPase cytoplasmique
Drg1 était essentielle à la maturation des grandes sous-unités ribosomiques et au recyclage d’un nouveau facteur
navette, la GTPase Nog1, que l’on pensait jusque là exclusivement nucléaire. L’activité d’ATPase de Drg1 est
nécessaire à sa fonction. Elle intervient très rapidement après l’export des particules, et en amont de Rei1,
définissant une nouvelle étape dans la maturation des ribosomes eucaryotes.
|
|
|
Conclusion |
|
Chez les eucaryotes, la maturation des sous-unités ribosomiques est couplée à leur transport entre les
différents compartiments cellulaires, et nécessite une régulation fine, aussi bien quantitative que qualitative. Mes
travaux de thèse ont permis, d’une part, d’affiner les connaissances sur le scénario d’assemblage des particules,
et d’autre part, de préciser le rôle de facteurs pré-ribosomiques impliqués à deux points majeurs de contrôle de la
biogenèse de la grande sous-unité, l’un dans le noyau, l’autre dans le cytoplasme.
De nombreuses questions restaient ouvertes concernant les mécanismes moléculaires régulant cette voie
métabolique complexe. En particulier, la nature du signal permettant la discrimination entre les particules préribosomiques
et les sous-unités ribosomiques matures est encore hypothétique. Le facteur Tif6, qui peut inhiber
l’association entre les sous-unités 40S et 60S, est un bon candidat pour jouer ce rôle, mais le mécanisme
moléculaire réel des dernières étapes de la biogenèse restait à établir.
L’un des principaux écueils rencontrés lors de l’étude de
l’assemblage du ribosome eucaryote a longtemps été l’absence d’informations
structurales concernant le ribosome mature. Afin de comprendre comment les protéines ribosomiques
s’enchâssent les unes par rapport aux autres, nous avons longtemps raisonné en nous basant sur
la structure connue du ribosome de l’archae Thermus thermophilus (Yusupov et al., Science 2001). Ceci
nous a régulièrement conduits à des hypothèses erronées, et s’avère
insuffisant pour l’étude des régions du ribosome spécifiques des eucaryotes. En 2010, une
avancée majeure dans le domaine a été la publication de la première structure du ribosome de
levure (Ben-Shem et al., Science 2010). Elle fut suivie de plusieurs structures raffinées, ou de complexes
avec d’autres protéines.
Plusieurs études structurales ont permis
d’obtenir la structure des particules pré-60S tardives, encore liées aux facteurs Arx1, Alb1,
Tif6, Rei1 et Jjj1 (Gartmann et al., J. Biol. Chem. 2010; Klinge et al., Science 2011;
Bradatsch et al., Nat. Struct. Mol. Biol. 2012; Greber et al., Nat. Struct. Mol. Biol. 2012,
Cell 2015). Le dimère Arx1-Alb1 est positionné à proximité immédiate du canal de sortie du peptide
naissant, confirmant une hypothèse reposant sur des données stériques, et sur
l’homologie d’Arx1 avec des protéines de la famille des méthionine-aminopeptidases
(Hung et Johnson, Mol. Cell. Biol. 2006). Cette position bloquerait le recrutement de certains facteurs de traduction tant
que la particule est immature. Rei1 est localisé à proximité immédiate. Jjj1, un autre facteur
caractérisé par l’équipe de Micheline Fromont-Racine (Demoinet et al., RNA 2007) constitue
l’interface entre Rei1 et Arx1. Tif6 occupe l’interface entre la petite et la grande
sous-unité ribosomique, et bloque donc l’interaction entre particules immatures, comme les
études fonctionnelles précédentes le laissaient supposer.
|
|
|
|



